JAB-HCCのshinyアプリ
2015年、虎の門病院肝臓センターからこんな報告がありました。
本邦における B 型慢性肝疾患からの肝発癌予測リスクスコアモデル(Japanese risk estimations of HBV-related HCC:JAB-HCC)の作成
https://www.jstage.jst.go.jp/article/kanzo/56/9/56_477/_pdf
Cox 比例ハザードモデルを用いて、リスクスコアモデルを作成したというお話です。抗ウイルス療法無治療のB型慢性肝疾患症例1143例で検討されています。これだけの症例をしっかりデータ取れるのは、さすが虎ノ門です。古い症例はHBV-DNA測定系が違うはずですが、保存血清で全症例に現行のリアルタイムPCR方で再測定した結果を用いているとのこと…羨ましいかぎりです。
肝発癌に寄与する因子として抽出された,年齢,性別,肝硬変の有無,ALT 値,
AFP 値,血小板数,HBeAg の有無,HBVDNA 量の計8 因子は、各項目の重み付けを行い、点数の総和を検査するだけで、リスクを判定することができます。
患者さんに説明する際にも、核酸アナログを内服するリスク、内服しないリスクを説明しやすくなりとても重宝しておりました。たしかどこかの製薬会社のパッフレットにも早見表のようなものがあった気がします。ところが先日患者さんに説明しようとした際に、あれ!スコア表が無いぞ…汗
文献が見つかったので良かったのですが、資料を探す暇もおしいので、shinyアプリにしておきました。当院はshiny-severにアップしてあるので、ブラウザのお気に入りから、すぐ使えます。
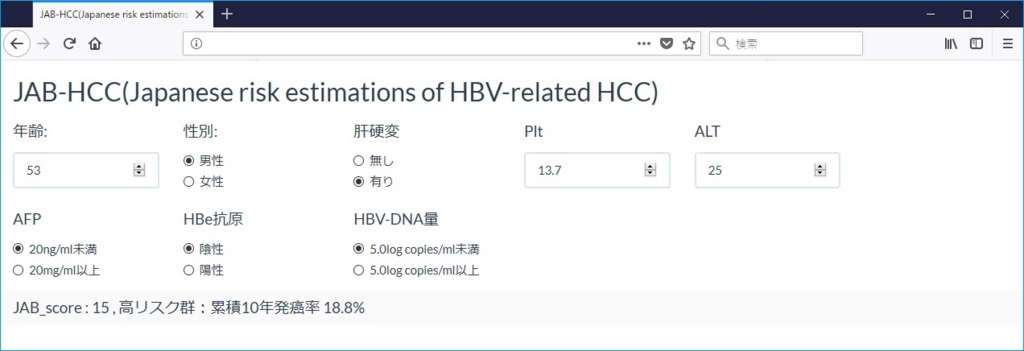
shinyを勉強するのに参考になればと思い、汚いコードですが晒しておきます。すこし直せばいろいろなスコア計算のアプリができるので、よく使うものは自分用にスコア計算アプリを作ってみてはどうでしょうか。
library(shiny)
library(shinythemes)
ui <- fluidPage(theme = shinytheme("flatly"),
titlePanel("JAB-HCC(Japanese risk estimations of HBV-related HCC)"),
fluidRow(
column(2,
numericInput("age",
label = h4("年齢:"),
value = 50)
),
column(2,
radioButtons("gender",
label = h4("性別:"),
c("男性" = "1","女性" = "2"))
),
column(2,
radioButtons("LC_JAB",
label = h4("肝硬変"),
choices = list("無し" = 0, "有り" = 4),
selected = 0)
),
column(2,numericInput("Plt",
label = h4("Plt"),
value = 25,
step=1)
),
column(2,numericInput("ALT",
label = h4("ALT"),
value = 35,
step=1)
)
),
fluidRow(
column(2,
radioButtons("AFP_JAB",
label = h4("AFP"),
choices = list("20ng/ml未満" = 0, "20mg/ml以上" = 2),
selected = 0)
),
column(2,
radioButtons("HBeAg_JAB",
label = h4("HBe抗原"),
choices = list("陰性" = 0, "陽性" = 3),
selected = 0)
),
column(3,
radioButtons("HBVDNA_JAB",
label = h4("HBV-DNA量"),
choices = list("5.0log copies/ml未満" = 0, "5.0log copies/ml以上" = 2),
selected = 0)
)
),
fluidRow(style="background-color:#fafafa;",
column(12,
h4(textOutput("text_JABHCC"))
)
)
)
server <- function(input, output) {
output$text_JABHCC <- renderText({
gender <- as.numeric(input$gender)
if(gender == 1){
genderJAB <- 4 # male
} else if(gender == 2){
genderJAB <- 0 #female
}
age <- as.numeric(input$age)
if(age <= 44){
ageJAB <- 0
} else if(age >= 45 & age <= 49){
ageJAB <- 3
} else if(age >= 50 & age <= 54){
ageJAB <- 5
} else if(age >= 55){
ageJAB <- 6
}
LCJAB <- as.numeric(input$LC_JAB)
ALT <- as.numeric(input$ALT)
if(ALT < 45){
ALTJAB <- 0
} else if(ALT >= 45){
ALTJAB <- 1
}
AFPJAB <- as.numeric(input$AFP_JAB)
Plt <- as.numeric(input$Plt)
if(Plt >= 15){
PltJAB <- 0
} else if(Plt < 15){
PltJAB <- 2
}
HBeAgJAB <- as.numeric(input$HBeAg_JAB)
HBVDNAJAB <- as.numeric(input$HBVDNA_JAB)
JAB_score <- genderJAB + ageJAB + LCJAB + ALTJAB + AFPJAB + PltJAB + HBeAgJAB + HBVDNAJAB
if(JAB_score <= 6){
comment_JAB <- c("低リスク群:累積10年発癌率 0.6%")
} else if(JAB_score >= 7 & JAB_score <= 10){
comment_JAB <- c("中リスク群:累積10年発癌率 2.2%")
} else if(JAB_score >= 11 & JAB_score <= 15){
comment_JAB <- c("高リスク群:累積10年発癌率 18.8%")
} else if(JAB_score >= 16 & JAB_score <= 24){
comment_JAB <- c("超高リスク群:累積10年発癌率 61.5%")
}
paste("JAB_score :", JAB_score,", ", comment_JAB)
})
}
shinyApp(ui = ui, server = server)